L’EFSA a publié en mai 2022 un article relatif au projet « Analyse du microbiote pour l’évaluation de risque : évaluation des substances alimentaires dangereuses et son rôle potentiel sur la variabilité et la dysbiose du microbiome intestinal » dans un numéro spécial consacré à l’évaluation européenne des risques alimentaires (EU-FORA).
Compte tenu du développement des domaines liés aux probiotiques et aux interventions ciblées sur le microbiome, il est nécessaire de spécifier clairement les critères et de fournir des détails sur les moyens et les approches pour atteindre ces critères avec pour objectifs que les fabricants puissent lancer de nouveaux produits et bénéficier d’une manière transparente de communiquer la qualité du produit aux utilisateurs finaux.
Le projet proposé « Analyse du microbiote pour l’amélioration de l’évaluation des risques : évaluation des substances alimentaires dangereuses et son rôle potentiel sur la variabilité et la dysbiose du microbiome intestinal » a été développé au sein de la Faculté de pharmacie de l’Université de Grenade (UGR) et de l’Institut « Jose Mataix Verdu » of Nutrition and Food Technology (INYTA – UGR), équipes de recherche qui effectuent l’analyse du microbiote à différentes fins de santé depuis 2003. L’intérêt actuel des déterminations du microbiote intestinal pour compléter l’évaluation des risques de traces de métabolites de substances toxiques et xénobiotiques dans les aliments est d’une grande pertinence . Dans le cadre de cette initiative, le consortium multidisciplinaire a soumis la proposition suivante de subvention de partenariat de l’EFSA (2019-2021) qui a été évaluée et récompensée avec succès : « KNOWLEDGE PLATFORM FOR ASSESSING THE RISK OF BISPHENOLS ON GUT MICROBIOTA AND ITS ROLE IN OBESOGÉNIC PHENOTYPE : LOOKING FOR BIOMARKERS » Acronyme : OBEMIRISK. Le programme a été supervisé par le Dr Margarita Aguilera-Gomez, professeure associée à l’UGR. Le programme consistait en trois modules indépendants différents basés sur les travaux de projet de recherche en cours et les intérêts de recherche antérieurs :
- Objectif/Module 1. Se concentrer sur l’obtention des informations les plus récentes sur la variabilité du microbiote humain et la dysbiose associée et/ou supposément causée par l’exposition et la consommation de substances dangereuses dans l’alimentation, et les perspectives futures des probiotiques et des probiotiques de nouvelle génération.
Une revue systématique, une méta-analyse et une méthodologie de recherche documentaire approfondie ont été décrites et appliquées pour obtenir des documents pertinents pour l’analyse holistique du microbiote humain et de son rôle dans l’évaluation des risques et pour élaborer des lignes directrices compilant des données réglementaires et des preuves scientifiques concernant le microbiote et les probiotiques, l’évaluation des risques et les aspects de sécurité alimentaire. La réglementation des probiotiques commercialisés diffère selon les pays. Ils peuvent être lancés comme des nutraceutiques ou des agents biothérapeutiques du vivant, des aliments médicaux, des produits biologiques ou des compléments alimentaires selon les pays. De plus, le niveau de classification de base n’est pas harmonisé à l’échelle mondiale.
- Objectif/Module 2. Apprendre les principales méthodes et technologies omiques disponibles pour l’analyse du microbiote intestinal (composition/modèles d’activité) tout en étant exposé à différents niveaux de substances dangereuses dans l’alimentation (par exemple, le bisphénol A et ses analogues).
- Objectif/Module 3. Connaître les principales méthodes de dosage chimique des bisphénols et analogues dans les échantillons alimentaires et humains (salive, urine, fèces) et élaboration de questionnaires communs et d’enquêtes pour l’estimation de l’exposition alimentaire à la présence de BPA et analogues ( conception, amélioration, validation et sa mise en œuvre).
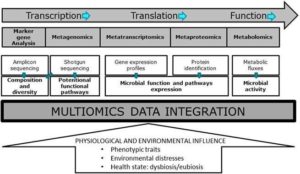 L’intégration de données multi-omiques est une mission innovante, en raison de la complexité et de la diversité accrues des données collectées. Cette intégration repose de plus en plus sur des outils bioinformatiques performants et des méthodes statistiques avancées. Par conséquent, l’intégration de données multi-omiques pose toujours des défis, mais l’intégration de plusieurs ensembles de données métaomiques présente une approche prometteuse pour caractériser de manière exhaustive la composition, l’activité fonctionnelle et métabolique des microbiomes. Ceci est particulièrement important pour que la recherche sur le microbiome soit traduite en applications cliniques. De plus, des études de cohorte prospectives importantes sont nécessaires pour valider les résultats et déterminer la reproductibilité des biomarqueurs pour améliorer davantage la gestion de la santé humaine (voir la figure ci-dessous, extraite de la revue EFSA).
L’intégration de données multi-omiques est une mission innovante, en raison de la complexité et de la diversité accrues des données collectées. Cette intégration repose de plus en plus sur des outils bioinformatiques performants et des méthodes statistiques avancées. Par conséquent, l’intégration de données multi-omiques pose toujours des défis, mais l’intégration de plusieurs ensembles de données métaomiques présente une approche prometteuse pour caractériser de manière exhaustive la composition, l’activité fonctionnelle et métabolique des microbiomes. Ceci est particulièrement important pour que la recherche sur le microbiome soit traduite en applications cliniques. De plus, des études de cohorte prospectives importantes sont nécessaires pour valider les résultats et déterminer la reproductibilité des biomarqueurs pour améliorer davantage la gestion de la santé humaine (voir la figure ci-dessous, extraite de la revue EFSA).
Le programme de recherche a réalisé la collecte et l’analyse d’échantillons fécaux d’enfants (n = 109), âgés de 3 à 13 ans, regroupés selon différents niveaux de mesure des bisphénols et de ses analogues afin d’identifier la composition du microbiote et les modèles de phénotypes de dysbiose. Les entérotypes du microbiote intestinal ont également été explorés. La collecte, la collation, la comparaison et l’intégration de données sur le microbiome contribuent à approfondir les connaissances sur le phénotype de la dysbiose-composé xénobiotique/toxique. Les résultats de ce programme de recherche seront publiés dans des publications en libre accès.
Pour en savoir plus sur ce projet, vous pouvez consulter ce lien.
Orchidali peut vous aider à commercialiser une souche probiotique.

Analyse du microbiote pour l’évaluation de risque
L’EFSA a publié en mai 2022 un article relatif au projet « Analyse du microbiote pour l’évaluation de risque : évaluation des substances alimentaires dangereuses et son rôle potentiel sur la variabilité et la dysbiose du microbiome intestinal » dans un numéro spécial consacré à l’évaluation européenne des risques alimentaires (EU-FORA).
Compte tenu du développement des domaines liés aux probiotiques et aux interventions ciblées sur le microbiome, il est nécessaire de spécifier clairement les critères et de fournir des détails sur les moyens et les approches pour atteindre ces critères avec pour objectifs que les fabricants puissent lancer de nouveaux produits et bénéficier d’une manière transparente de communiquer la qualité du produit aux utilisateurs finaux.
Le projet proposé « Analyse du microbiote pour l’amélioration de l’évaluation des risques : évaluation des substances alimentaires dangereuses et son rôle potentiel sur la variabilité et la dysbiose du microbiome intestinal » a été développé au sein de la Faculté de pharmacie de l’Université de Grenade (UGR) et de l’Institut « Jose Mataix Verdu » of Nutrition and Food Technology (INYTA – UGR), équipes de recherche qui effectuent l’analyse du microbiote à différentes fins de santé depuis 2003. L’intérêt actuel des déterminations du microbiote intestinal pour compléter l’évaluation des risques de traces de métabolites de substances toxiques et xénobiotiques dans les aliments est d’une grande pertinence . Dans le cadre de cette initiative, le consortium multidisciplinaire a soumis la proposition suivante de subvention de partenariat de l’EFSA (2019-2021) qui a été évaluée et récompensée avec succès : « KNOWLEDGE PLATFORM FOR ASSESSING THE RISK OF BISPHENOLS ON GUT MICROBIOTA AND ITS ROLE IN OBESOGÉNIC PHENOTYPE : LOOKING FOR BIOMARKERS » Acronyme : OBEMIRISK. Le programme a été supervisé par le Dr Margarita Aguilera-Gomez, professeure associée à l’UGR. Le programme consistait en trois modules indépendants différents basés sur les travaux de projet de recherche en cours et les intérêts de recherche antérieurs :
Une revue systématique, une méta-analyse et une méthodologie de recherche documentaire approfondie ont été décrites et appliquées pour obtenir des documents pertinents pour l’analyse holistique du microbiote humain et de son rôle dans l’évaluation des risques et pour élaborer des lignes directrices compilant des données réglementaires et des preuves scientifiques concernant le microbiote et les probiotiques, l’évaluation des risques et les aspects de sécurité alimentaire. La réglementation des probiotiques commercialisés diffère selon les pays. Ils peuvent être lancés comme des nutraceutiques ou des agents biothérapeutiques du vivant, des aliments médicaux, des produits biologiques ou des compléments alimentaires selon les pays. De plus, le niveau de classification de base n’est pas harmonisé à l’échelle mondiale.
Le programme de recherche a réalisé la collecte et l’analyse d’échantillons fécaux d’enfants (n = 109), âgés de 3 à 13 ans, regroupés selon différents niveaux de mesure des bisphénols et de ses analogues afin d’identifier la composition du microbiote et les modèles de phénotypes de dysbiose. Les entérotypes du microbiote intestinal ont également été explorés. La collecte, la collation, la comparaison et l’intégration de données sur le microbiome contribuent à approfondir les connaissances sur le phénotype de la dysbiose-composé xénobiotique/toxique. Les résultats de ce programme de recherche seront publiés dans des publications en libre accès.
Pour en savoir plus sur ce projet, vous pouvez consulter ce lien.
Orchidali peut vous aider à commercialiser une souche probiotique.
Tags: